염기서열분석(DNA direct sequencing)은 염기서열 이상으로 발생된 유전질환을 진단하기 위한 표준검사로 분자진단검사에서는 핵심적인 진단기술이다. 1977년 Sanger에 의해 개발된 chain-termination method가 25년간 널리 사용된 염기서열분석법이었는데 이 기술은 최근에 더욱 빠른 속도로 발전하여 나노포어 염기서열분석(nanophore sequencing)과 같은 전혀 새로운 개념의 기술이 등장하고 있을 뿐만 아니라, 속도 측면에서도 2000년대 중반까지 약 30년 동안 이룬 발전보다 최근 5년간 발전이 이를 훨씬 앞지르고 있다.
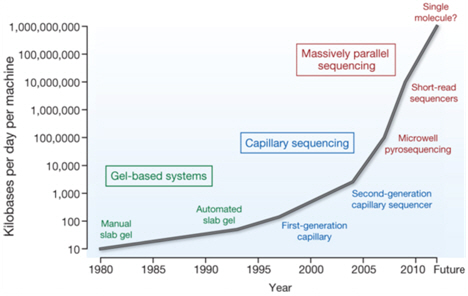
이러한 기술적 발전으로 인해 지금껏 염기서열분석의 임상 이용을 제한하던 시간 및 비용 문제가 해결될 조짐을 보이면서 분자진단검사도 새로운 전환기를 맞고 있다. 예컨대, 유전자검사(genetic testing), 즉 개별유전자에 대한 접근법(gene by gene approach)이 현재 분자진단검사의 기본전략이었다면 앞으로는 다양한 질환에서 유전체검사(genomic testing), 즉 전체 유전체에 대한 접근법(genomic approach)이 기본전략이 될 가능성이 높다. 여기서는 염기서열분석법의 기술적 발전과 유전질환에서 이의 임상적 이용 및 유용성에 대해 개괄적인 내용을 알아보고자 한다.
2. 차세대 염기서열 분석기법의 특징과 데이터
(1) 차세대 염기서열 분석기법(NGS technology)
2007년 Roche가 처음으로 NGS 장비를 출시한 이후, lllumina Solexa, Applied Biosystems 등이 연이어 NGS 장비를 소개하였다. 현재의 Sanger 염기서열분석장비는 기껏해서 동시에 수십 개의 전기영동이 가능한 반면, NGS 염기서열분석장비에서는 수십만 내지 수십억 개의 서로 다른 염기서열분석 반응이 동시에 진행되고 판독된다. 장비마다 차이는 있으나 NGS 염기서열분석 과정은 검체 준비, 클론성 증폭, 염기서열분석의 3단계로 구성된다.
1) 검체 준비sample preparation step): 검체는 분석 대상-유전체 전체,엑솜, 특정 염색체 부위, 특정 유전자 조합
에 따라 분절법, 포획법 또는 증폭법 등으로 준비된다. DNA 절편은 장비에 따라 읽기 적절한 길이(bp)로 준비되고, 여기에 동시 증폭 및 염기서열반응을 위한 adaptor가 결합된다.
2) 클론성 증폭(clonal amplification step): 준비된 절편을 플레이트 위 일부 공간 또는 droplet 안에서 클론으로 증폭시킨다.
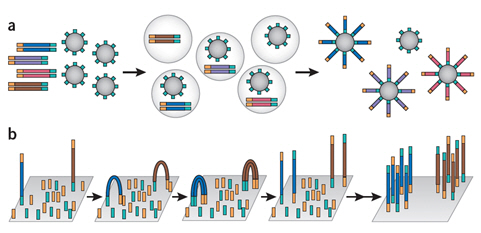
3) 염기서열분석(sequencing reaction step): 클론성 증폭이 이루어진 수십만 내지 수십억 개의 반응산물의 염기서열을 동시에 분석한다. 위 과정은 장비에 따라 1일~1주가 소요되며, 한 번의 분석과정에서 최대 600 Gb의 염기서열 정보(참고: 인간의 전체 유전체 길이는 3 Gb이며, 2003년에 완결된 인간유전체 프로젝트의 경우 10년 이상의 기간에, 100억달러가 소요되었다)를 얻을 수 있다. NGS는 대용량의 정보를 제공할 수 있으므로, 전체 유전체 외에도 엑솜, 특정 유전자 조합 또는 특정 염색체 부위의 염기서열정보를 얻는데 사용될 수 있다. 또한 minor allele을 동시에 분석할 수 있고, 상대적 정량도 가능하므로, 미생물이나 종양처럼 기원이 다른 핵산이 혼합된 경우의 정량적 분석에도 사용될 수 있다.
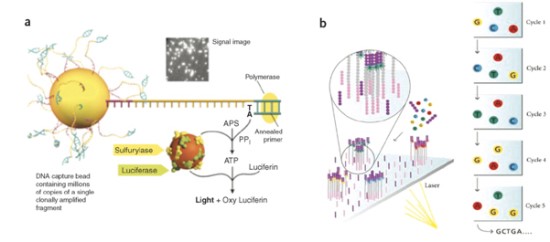
(2) NGS 데이타의 특징
NGS 데이터가 Sanger 염기서열분석 데이터와 다른 몇 가지 특징이 있는데, 첫번째는 외가닥 염기서열분석(single strand sequencing)이다. Sanger 염기서열분석 데이터는 sequencing reaction에 사용된 모든 세포의 DNA 염기서열의 총합으로만 표현되는 반면, NGS 데이터는 각 세포에서 유래한 외가닥 DNA 염기서열이 각각 독립적으로 표현된다. 이를 바꿔 말하면 NGS 데이터는 Sanger 데이터보다 polymerase error에 훨씬 취약하다는 약점이 있다. 따라서 NGS 데이터에서 특정 위치의 염기를 최소 몇 번 읽었는지(coverage depth), 염기변이의 빈도가 충분히 높은지 중요한 이유가 바로 이것에 있다.
두번째, NGS는 대용량 데이터(mass data)이다. 엑솜만 하더라도 보통 수 Gb의 텍스트 파일을 다루게 된다. 따라서 Sanger 데이터와 비교가 어려울 정도로 대용량의 정보를 얻을 수 있지만, 이로 인해 분석 복잡도가 크게 증가하여 고사양의 전산 자원과 보다 빠른 속도의 분석 알고리즘이 필요하다.
세번째, NGS의 기본 데이터 유형은 텍스트파일이다. Sanger에서는 전기영동 내내 시간에 따른 형광 종류별 강도 변화가 기록된 electropherogram이 주어지므로, base calling 과정을 거친 텍스트 파일 외 각 peak 높이, peak간 간격, peak의 상대적 비율과 변화 등 다양한 정보를 통해 sequencing quality 및 분석소프트웨어의 분석 오류에 대한 직관적 파악이 가능하다. NGS도 장비에서 기록된 신호 데이터가 있지만, 매 사이클마다 한 번씩 기록된 형광이고 이는 base calling 과정을 거친 텍스트 파일과 본질적으로 다르지 않다. 따라서 NGS 데이터는 sequencing quality에 대한 직관적 파악이 쉽지 않고, 분석소프트웨어의 분석 오류 발생시 이를 파악하기가 어렵다.
3. NGS의 분자진단에서 고려할 사항
1) 검체 준비
첫번째, 분석대상의 크기 및 수에 따라 적절한 방법(증폭법, 캡춰법)을 선택하여야 한다. 증폭법은 설계가 간단하고 개개 분석대상에 따라 최적화된 조건을 잡는 것이 용이하지만 분석대상이 많아지면 시발체 합성 비용이 이에 따라 증가하므로 분석대상의 크기가 비교적 작을 때 유리하다. 반면, 캡춰법은GC가 많은 부위를 놓칠 수 있지만 분석대상을 확장하는 것이 용이하고 유전자당 비용이 매우 낮아 분석대상의 크기가 클 때 유리하다. 두번째, 특정 유전자 또는 특정 염색체 부위를 선택적으로 분석하는 경우에 해당 부위가 제대로 캡춰 또는 증폭되는지 확인하는 것이 필요하다. 엑솜 캡춰의 경우 사용하는 시스템에 따라 5% 내외의 유전자 부위가 캡춰되지 않기도 한다. 세번째, 검체 준비는 수작업인 경우가 많고 다수의 복잡한 단계를 거치므로 이에 대한 검증 및 정도관리가 필요하다.
2) NGS 염기서열분석장비
NGS 장비의 처리량과 검체당 비용을 고려하여 목적에 맞는 것을 선택할 필요가 있다. 분석 목적이 엑솜 내지 전체 유전체라면 적어도 수십 Gb의 처리량을 갖는 장비가 필요하지만, 수십 종 내지 수백 종의 유전자를 분석하는 다중유전자패널이라면 처리량이 수십 Mb ~ 수 Gb인 장비가 적당하다. 또한 NGS 장비는 염기서열분석에 사용하는 기술이 서로 상이하고 이에 따라 취약한 염기서열분석 오류가 다를 수 있다. 예를 들어, Roche 장비는 homopolymer error에 좀 더 취약하고, Illumina는 한 번에 읽을 수 있는 절편의 길이가 작으므로 misalignment에 기인한 오류에 취약한 것으로 알려져 있다.
3) NGS 데이터 분석 및 해석
NGS 염기서열분석을 통해 발견되는 코딩 부위 및 인접 인트론 부위의 염기변이는 때때로 유전자당 수십 개에 이르지만 이 중 상당수는 sequencing error 또는 misalignment error 등에 기인한 것이다. 따라서 염기변이의 임상적 해석 이전에 이것이 실재하는 염기변이인지 Sanger sequencing등으로 확인하기 위한 과정이 필수적이다. 실재하는 염기변이임이 확인되었다면, 이의 임상적 의미를 해석하기 위해서 해당 염기변이의 정상인 빈도, 해당 아미노산 종간 보존 여부 및 구조적 기능적 중요도, 가족에서 segregation analysis 등의 방법을 이용한다. 때에 따라 PolyPhen, SIFT 등의 in-silico prediction software의 정보를 이용할 수도 있다.
4. 결론
NGS 기술은 지금까지 가능하지 않았던 새로운 유형의 분자진단검사를 가능하게 해줄 것으로 기대된다. 진단 및 예후 판정에 수십 종의 유전자발현 프로필이 이용되는 암과 같은 복합유전질환, 최소 수십-수백 개의 원인유전자에 의해 발병하는 단일유전질환의 분자진단 등이 바로 그것이다. 따라서 지금은 당연한 것으로 여겨지는 진단 알고리즘과 검사들 중 상당수가 이로 인해 바뀔 가능성이 높다.
미국 Emory 대학병원의 유전검사실이 개설한 congenital disorder of glycosylation panels, congenital muscular dystrophy panels, X-linked intellectual disability panels의 유전자검사패널은 새로운 분자진단검사의 좋은 예이다. 또한 국내에서도 질병관리본부가 지원하는 희귀질환 유전자진단 지원사업에서 familial hypertrophic or dilated cardiomyopathy, fanconi anemia, limb-girdle muscular dystrophy 등의 유전자진단에 NGS 기반의 유전자검사패널이 이미 이용되고 있다. 진단목적의 검사로 당장 상용화하기에 기술적, 분석적 어려움이 존재함에도 불구하고 NGS 기술은 조만간 분자진단검사실에서 가장 중요한 검사기술이 될 것으로 전망된다.
참고 서울의대 성문우의 학회 발표자료
