연세대학교 의과대학 약리학교실 김형범 교수팀

연세대학교 의과대학 약리학교실 김형범 교수와 서상연 연구원 연구팀은 생체 내 전달에 유리한 소형 유전자가위를 선별할 수 있는 인공지능 기반 모델을 개발했다고 밝혔다.
유전자가위는 타깃 DNA를 손쉽게 바꿔줄 수 있는 특성이 있어 유전자 치료 분야에서 널리 사용되며 이러한 유전자가위를 각 신체 부위로 이동시키는 데는 아데노부속바이러스가 많이 사용되는데 SpCas9과 같은 전자가위는 상대적으로 큰 단백질이기 때문에 아데노부속바이러스를 통한 전달이 불리한 면이 있다.
따라서, 그동안 다양한 연구에서 Cas9와 같은 소형 유전자가위들이 개발돼 왔으며 연구팀은 소형 유전자가위의 사용에 있어 유용한 가이드를 제시하고자 선별모델을 개발하기 위한 연구를 진행했다.
연구팀은 먼저 다양한 소형 유전자가위 중 유전자 연구에 응용 가능성이 높은 소형 유전자가위(Cas9)들을 선별하고 총 17개의 Cas9의 활성도와 특이도를 수만개의 표적·비표적 DNA에서 측정하고 이를 비교분석했다.
유전자가위의 활성도는 표적으로 하는 DNA를 얼마나 잘 절단하는지 보여주는 지표고, 특이도는 해당 유전자가위가 얼마나 부작용(원하지 않는 DNA의 절단)이 적은지를 보여주는 지표다.
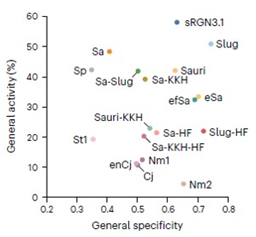
분석 결과, 기존의 널리 사용되고 있던 유전자가위 SpCas9보다 크기는 작으면서 활성도와 특이도가 높은 2개의 소형 Cas9(sRGN3.1, SlugCas9)을 확인했다. SpCas9의 평균 활성도와 특이도는 각각 42%, 0.35인데 반해, sRGN3.1은 각각 58%, 0.63, SlugCas9은 각각 51%, 0.74로 더 높게 나타났다.
이어서 분석 데이터를 바탕으로 인공지능을 이용해 소형 Cas9의 활성도와 특이도를 예측할 수 있는 인공지능 모델‘DeepSmallCas9’을 개발하고 유용성을 검증했다.
연구팀이 개발한 DeepSmallCas9은 소형 유전자가위의 활성도와 특이도를 동시에 검증할 수 있다. 기존 유전자가위 활성 예측 모델들은 활성도와 특이도 중 하나만을 검증할 수 있어 각각을 다른 모델을 이용해 예측해야하는 번거로움이 있었다.
해당 모델을 통해 분석을 진행했을 때 실제 임상에서 적용할 수 있는 유전자가위 선별에 유용함을 보여줬다.
실제 미국 국립생물공학정보센터의 ClinVar 데이터베이스에 공개된 1만 3145개의 우성 단일염기변이에 해당 모델을 적용해 본 결과, 82%(1만 844개) 이상의 돌연변이 DNA를 효율적으로 제거할 수 있는 유전자가위를 선별할 수 있는 것을 확인했다.
김형범 교수는 “이번 연구를 통해 소형 Cas9 대량 검증을 진행함으로써 기존에 사용되던 유전자가위보다 더 효과적인 소형 유전자가위들을 발견할 수 있었다”면서 “새롭게 개발한 인공지능 기반의 유전자가위 선별 모델 또한 통해 추후 기초연구, 유전자 치료제 개발의 속도와 효율을 높일 수 있을 것으로 기대한다”고 말했다.

이번 연구 결과는 국제학술지 ‘Nature Methods, (IF 47.99)'에 게재됐다.
