광주과학기술원(GIST) 전기전자컴퓨터공학부 남호정 교수 연구팀이 신규 약물 구조를 생성해주는 인공지능 모델을 개발했다고 밝혔다.

약물 효과를 최적화한 저분자 화합물 생성을 통해 신약 개발 시간을 획기적으로 단축하는 데 기여할 것으로 기대된다.
전통적인 신약개발 과정에서 선도물질의 최적화 단계는 수개월부터 수년까지 걸릴 수 있는 연구 과정을 필요로 하지만 인공지능을 도입하면 개발 시간을 몇 주에서 몇 개월로 크게 단축시킬 수 있다.
특히 이번에 개발된 인공지능 모델은 다양한 치료제 개발에 적용될 수 있도록 표적 단백질 정보를 쉽게 변경 가능토록 설계, 약물 개발 시장에 범용적으로 적용할 수 있는 강점을 갖고 있다.
연구팀은 기존의 전이 학습 모델의 문제점을 해결하기 위해 미세조정 단계에서 경험 기억 메모리(Experience memory)와 토너먼트 선택(Tournament selection)을 이용하는 방식을 고안해 생성모델이 더욱 다양한 화합물 구조를 탐색할 수 있는 훈련 알고리즘을 제안했다.
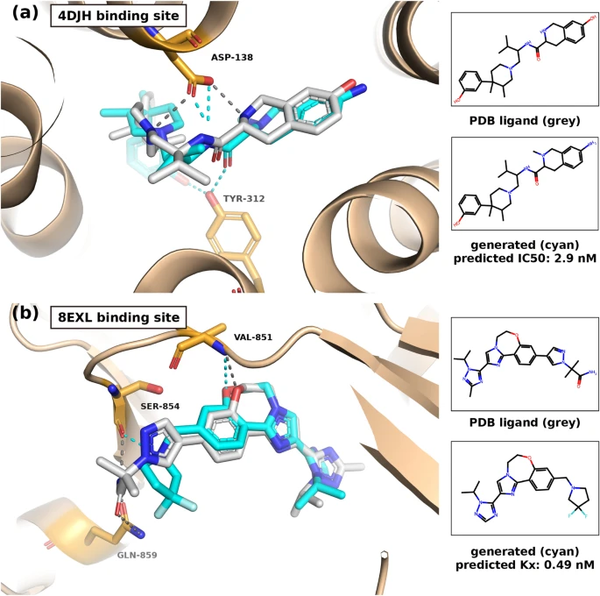
연구팀이 개발한 인공지능 모델이 제시한 분자 구조를 분석한 결과 약물 후보로 예측된 다수의 생성물이 기존의 데이터에 있는 분자와는 유사성이 낮은 신규 구조임을 밝혔다.
또 기존 화합물의 주요 골격은 유지하면서 세부 구조의 변화를 통해 표적 활성도 및 표적 단백질과의 3차원 구조적 결합을 향상하는 약물 최적화 방식도 적용 가능할 것으로 확인됐다.
남호정 교수는 "이번 연구 성과는 기존 전이학습 모델의 문제점을 해결해 안정적인 학습을 유도할 수 있고 데이터가 한정된 상황에서도 고품질의 다양한 신규 분자 구조를 제안하는 것이 가능하다"며 "신약 개발 초기 단계에 적용해 후보물질 및 선도물질 발굴 과정을 획기적으로 단축하고 효율성을 높일 수 있을 것으로 기대한다"고 말했다.

이번 연구결과는 화학정보학 분야 국제학술지 'Journal of Cheminformatics'에 게재됐다.
