고려대학교 융합생명공학과 구만복 교수 연구팀이 바이오센서에서 항체 대체로 사용되는 바이오리셉터인 앱타머를 고성능으로 최적화하는 방법을 개발했다.

앱타머는 단일 가닥의 핵산(DNA 또는 RNA)으로, 표적 물질에 특이적으로 검출하는 데에 사용되는 바이오리셉터로 1차 개발 과정인 셀렉스(SELEX; Systemic evolution of ligands by exponential enrichment)를 통해 얻은 앱타머가 결합 필수 서열 또는 구조 이외의 불필요한 부분을 가질 때 앱타머가 최상의 성능을 발휘하지 못하므로, 이후 다양한 후속개발과정(Post-SELEX optimization)이 진행된다.
연구팀은 이러한 후속 개발 과정에서 앱타머를 고성능화하는 최적화의 일환으로, 앱타머를 표적 물질에 결합한 채로 결합하지 않은 부위를 핵산 외부 가수분해 효소(Exonuclease) 2종으로 양방향에서 잘라내는 ‘nonessentials-guided in situ truncation’ 방법을 최초로 개발, 성공적으로 적용했다.
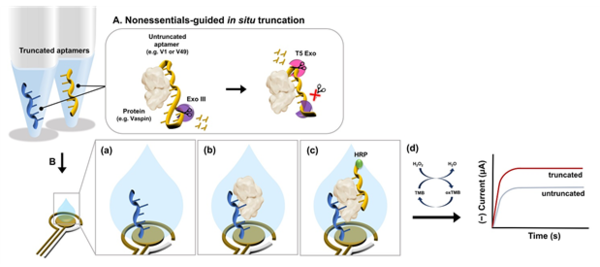
또한 해당 방법을 쌍 앱타머(Aptamer pair)에 적용한 결과, 표적 물질 결합에 중요한 부위만 남은 최적화된 길이의 앱타머를 얻을 수 있었고, 이는 매우 민감하고 특이적으로 표적 물질을 검출해 내는 것을 확인하였다.
연구팀은 이러한 고성능의 앱타머를 바이오센서에 이용함으로써, 바이오센서의 검출 성능을 향상할 수 있음을 알 수 있었다.

한국연구재단 기초연구과제의 지원으로 수행한 이번 연구결과는 ‘Chemical Engineering Journal’에 게재됐다.
